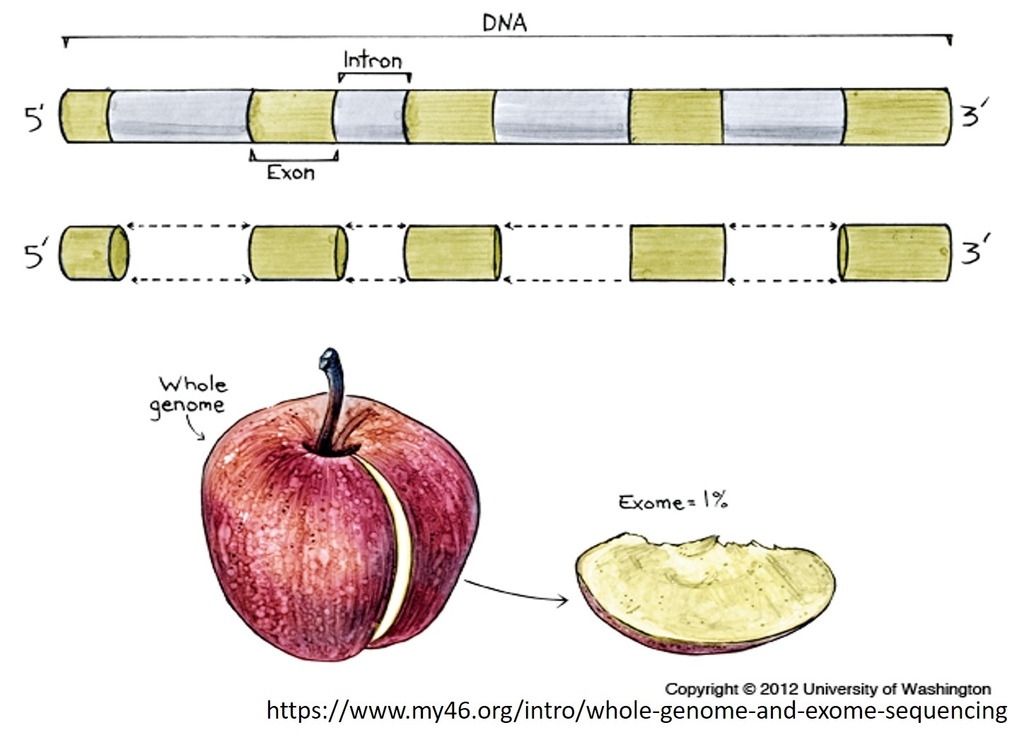
全外顯子定序(whole exome sequence, WES)在臨床上能夠快速作為單基因突變所導致的疾病檢測,同時在研究領域中更被用於探討多基因突變的複雜疾病,像是腫瘤、糖尿病及高血壓等。美國國家衛生研究院(NIH)心肺血液研究單位更是利用外顯子定序計畫(Exome Sequencing Project,ESP)探討基因與心肺及心血管相關疾病間的關聯性、功能及機制。另外,美國癌症基因體圖譜計畫TCGA (The Cancer Genome Atlas )與國際癌症基因組聯盟ICGC (International Cancer Genome Consortium)也都透過外顯子定序等定序技術及生物資訊分析,探討癌症相關致病基因及位點的變化[1]。
那什麼是全外顯子定序(whole exome sequence, WES)?
顧名思義就是將全部的外顯子做定序,那為什麼要這麼做呢?人類基因體(Genome)大約有30億個鹼基對,總共約有23000個基因,而其能轉譯成蛋白質的外顯子(exome)含有18萬個,共有3000萬個鹼基對占人體全部鹼基對的1%,目前科學證實,大多數DNA突變引起的疾病(約有85%)都發生在外顯子區域的序列變異[2,3],因此全外顯子定序(whole exome sequence, WES)相對於全基因組定序(whole genome sequence, WGS)雖然不能完整得到基因資訊,但卻更加經濟及高檢測覆蓋率[4]。

圖一、基因突變會導致疾病發生
外顯子定序能檢測不同的外顯子組序列變異,包含:單核甘酸變異(single nucleotide variants, SNVs)、插入或刪除短片段變異(insert or delete short segment, InDels)、及拷貝數變異(copy number variations, CNVs)等,同時搭配已知文獻中的生物資訊分析、線上資料庫工具做系統性地分析彙整,依序進行序列比對(Map)、序列異變分析(Variant Analysis)、探討可能的異變及其功能、重要性與生物意義等,提供一個完整的基因突變研究方法[4]。

圖二、台灣生物資訊核心設施(Taiwan Bioinformatics Institute)建立的外顯子組序列變異分析流程
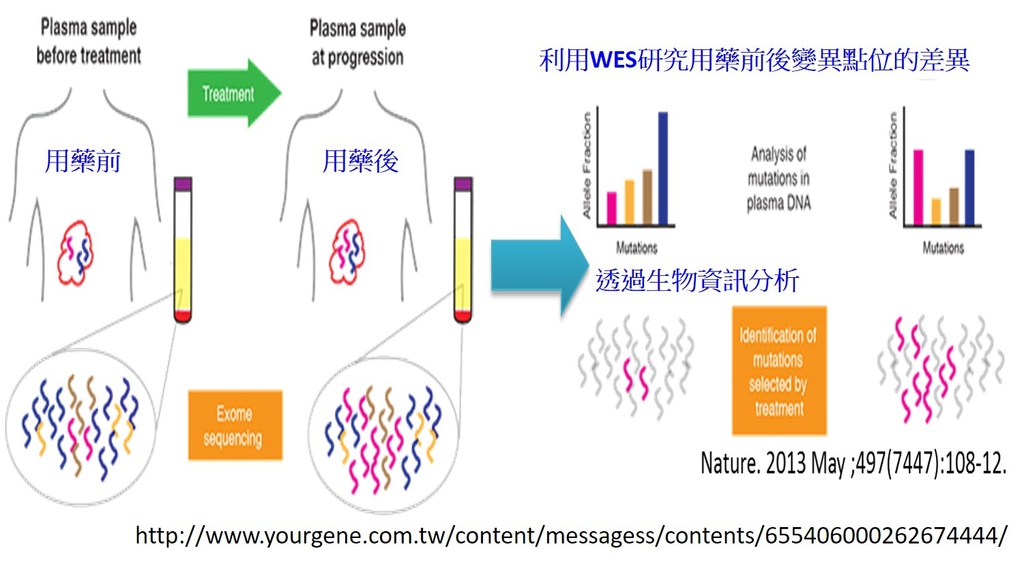
圖三、過去文獻中利用Exome Sequencing定序分析用藥前與用藥後基因變異點位的差異
全外顯子定序常見的應用,可用在癌症基因、遺傳性疾病、藥物敏感性、飲食、過敏、慢性病、膚質等相關疾病的基因檢測,提供完整基因資訊及相關知識,讓檢測者能夠在疾病發生前提早做預防或準備,讓傷害降到最低,同時也幫助下一代更健康[5]。

圖四、全外顯子定序常見的應用
當然,全外顯子定序還是有他的缺點,如果當致病基因不在外顯子當中,或者是某基因序列被複製或刪除,但基因序列本身並沒有改變,這樣的情況也是無法偵測到[6]。所以如果是檢測已知的基因突變,全外顯子定序是一個既方便且經濟的檢測方式,但如果是要用於科學未知的研究,則需要搭配更多的配套措施,做反覆性的驗證才能確保實驗的真實性。
參考資料:
1. Yourgene Health http://t.cn/EyAyZpm
2. Ng SB et al. "Targeted capture and massively parallel sequencing of 12 human exomes". Nature 2009. 461 (7261): 272–276.
3. Choi M et al. "Genetic diagnosis by whole exome capture and massively parallel DNA sequencing". PNAS 2009. 106 (45): 19096–19101.
4. TBI台灣生物資訊核心http://www.tbi.org.tw/enews/Nvol09/index.html
5. 解讀基因股份有限公司http://t.cn/EywDJGV
6. 科學月刊社 http://scimonth.blogspot.com/2015/05/blog-post_22.html


 留言列表
留言列表

